| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
- pandas row 제거
- 모델 freeze
- Prompt Tuning for Graph Neural Networks
- 선형함수 딥러닝
- 비선형함수
- Does GNN Pretraining Help Molecular Representation?
- weight 일부 고정
- sktime
- layer 일부 freeze
- pandas 행 제거
- molecular representation
- pytorch 데이터셋 나누기
- EDA in python
- pandas 특정 조건 열 제거
- 경우의 수 파이썬
- 일부 레이어 고정
- 판다스 조건
- pandas
- Graph Theory
- pytorch dataset split
- 시계열 라이브러리
- pretraining
- EDA 추천 파이썬
- Skip connection
- sktime 예제
- python 경우의 수
- 비선형함수 딥러닝
- sktime tutorial
- sktime 튜토리얼
- pandas 조건
- Today
- Total
MoonNote
GeomGCL: Geometric Graph Contrastive Learning for Molecular Property Prediction 본문
GeomGCL: Geometric Graph Contrastive Learning for Molecular Property Prediction
Kisung Moon 2021. 12. 15. 12:073줄 요약
- 2D molecular graph와 3D graph를 활용하여 contrastive learning
- distance와 angle 정보를 2D, 3D 관점에서 모두 활용
- 기존 geometric GNNs들과 성능 차이가 꽤 많이 남(SGCN, MAT, DimeNet 등)
Methods
1. Geometric Embedding
- local distance(2D), 2D angle, global distance(3D), 3D angle을 구하기 위해 선행 연구에서 사용된 radial basis function(RBF)를 사용하였다.
2. Adaptive Geometric Message Passing
(i) Node -> Edge Message Passing

- edge를 구성하는 pairwise node의 embedding과 bond feature(2D), global distance(3D)[둘다 edge 정보]를 concat
(ii) Edge -> Edge Message Passing

- (2D) target edge의 embedding과 neighbors edge와의 2D angle embedding을 element-wise 연산 후 sum

- (3D) target edge의 embedding과 neighbors edge와의 3D angle embedding을 element-wise 연산 후 sum
- (3D) 여러 개의 angle domain을 적용한 결과를 concat후 hierachical aggregation
(iii) Edge -> Node Message Passing

- target node의 neighbors edge embedding과 distance embedding 각각을 element-wise 연산 후 sum
(iv) Node -> Graph Attentive Pooling
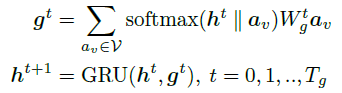
3. Geometric Contrastive Optimization


- local한 spatial correlation을 반영하기 위해 spatial regularizaion을 사용
- spatial loss + contrastive loss를 함께 사용

4. Downstream
- 2d encoder와 3d encoder의 결과를 sum

Result

- 기존 방법들과 꽤 많은 성능 차이가 남
원문
https://arxiv.org/abs/2109.11730



